Руководство администратора модуля управления Скала^р Геном версия 1.16#
1 Установка программы#
Для установки и работы ПО «Скала^р Геном» требуется операционная система Альт 8 СП релиз 9, Альт 8 СП релиз 10, Astra Linux Special Edition 1.7.3 (Орёл), RedOS 7.3.
1.1 Минимальные требования к устройству (виртуальной машине):#
- CPU: от 4 ядер;
- RAM: от 16 Гб;
- ROM: от 100 Гб SSD;
- NET: от 1 Гбит/с Ethernet.
1.2 Состав дистрибутива#
Дистрибутив содержит скрипт установки genome-installer.run.
1.3 Используемые сетевые порты#
Порты, необходимы для нормальной работы:
| ПOPT | ПРОТОКОЛ | НАПРАВЛЕНИЕ | НАЗНАЧЕНИЕ |
|---|---|---|---|
| 22 | SSH | I/O | SSH-подключение к модулю управления Геном |
| 5432 | TCP | I/O | PostgreSQL БД модуля управления Геном |
| 48800 | TCP | I/O | REST API модуля управления Геном |
| 50888 | TCP | I/O | Web-интерфейс модуля управления Геном |
| 15000-16000 | HTTP | I/O | IPMI proxies для управления серверами |
Дополнительные порты:
| ПOPT | ПРОТОКОЛ | НАПРАВЛЕНИЕ | НАЗНАЧЕНИЕ |
|---|---|---|---|
| 443 | HTTPS | I/O | Web-интерфейс с подключённым SSL-сертификатом |
1.4 Ход развёртывания#
1.4.1 Открыть терминал.
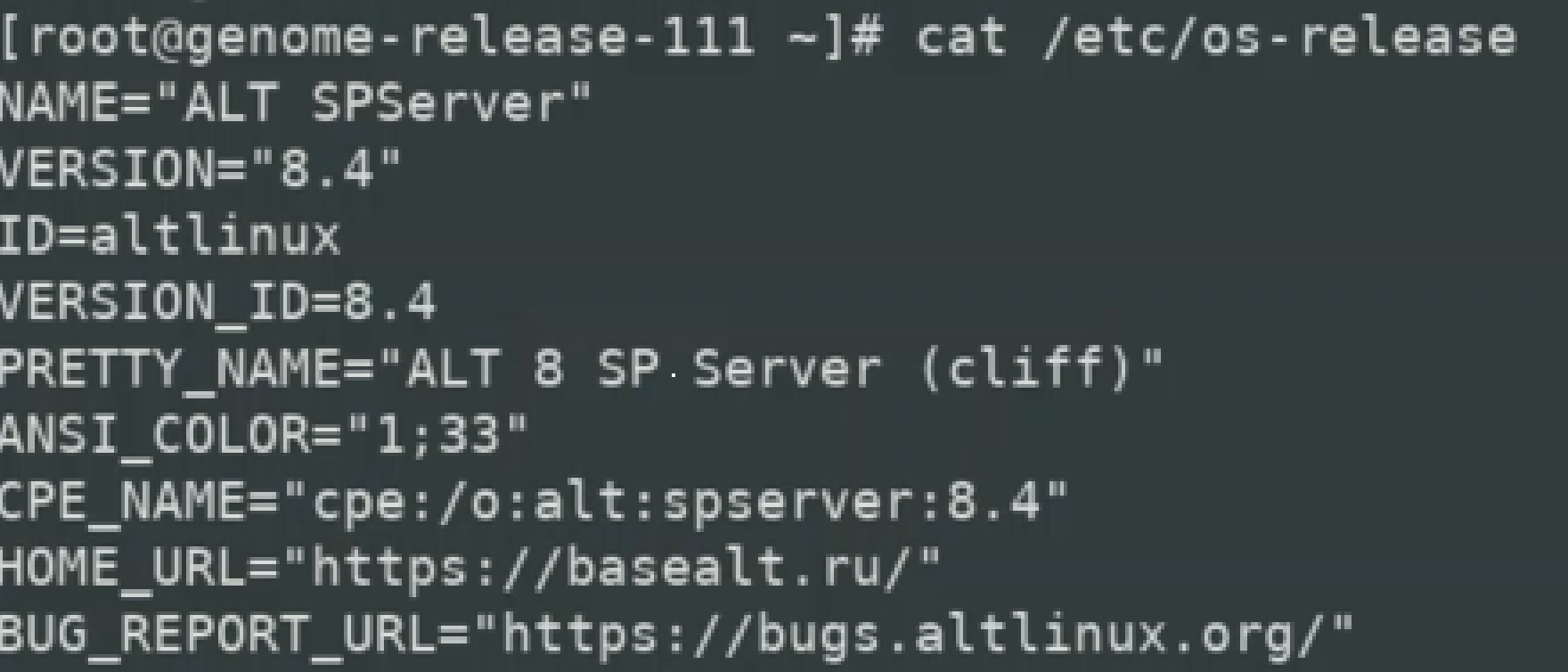
1.4.2 Проверить тип и версию установленной ОС:
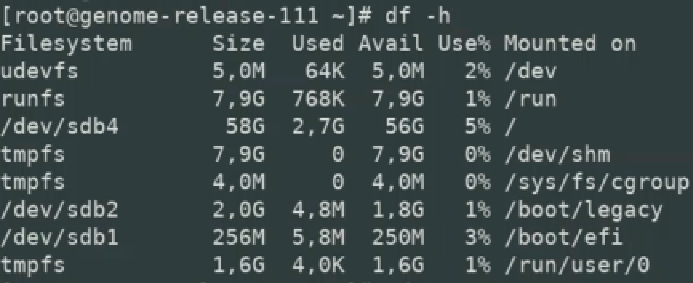
1.4.3 Проверить наличие свободного места для установки модуля управления ПО «Скала^р Геном»:
1.4.4 Перейти в корневой каталог:
1.4.5 Загрузить файл дистрибутива модуля управления ПО «Скала^р Геном» из репозитория.
Дождаться окончания загрузки:

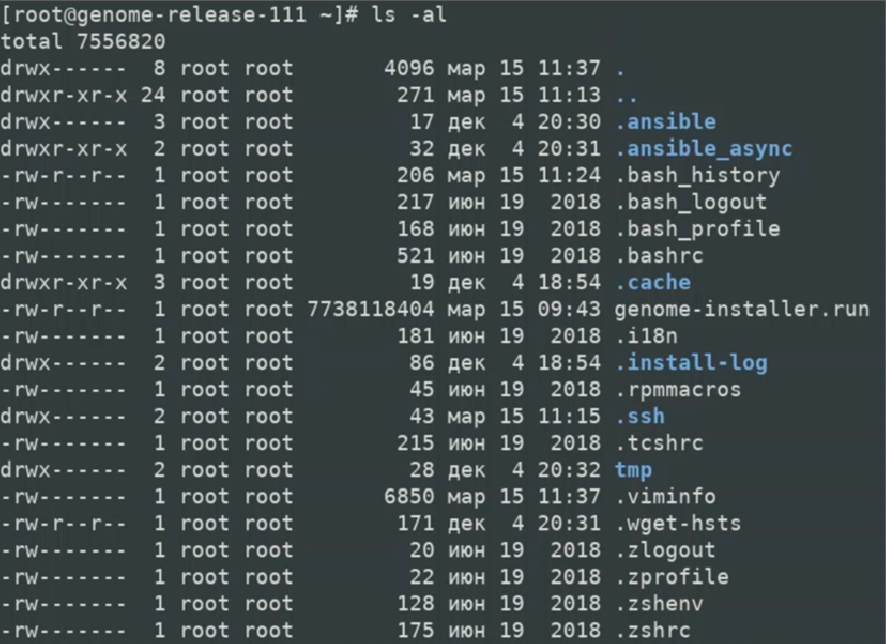
1.4.6 Вывести список файлов в директории и проконтролировать наличие файла genome-installer.run и прав доступа к нему:
1.4.7 Разрешить выполнение файла genome-installer.run:
1.4.8 Запустить выполнение файла genome-installer.run:
Начнётся процесс распаковки и установки модулей:
1.4.9 По завершении выполнения скрипта появится окно выбора опций, в котором выбрать нужную (здесь и далее, в случае необходимости, выбор подтверждается нажатием клавиши ПРОБЕЛ с последующим появлением символа «*» в соответствующем столбце):
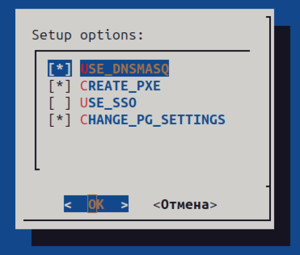
USE_DNSMASQ – настройки сервисов dnsmasq, таких как DNS, DHCP и tftp (не обязательно для Геном);
CREATE_PXE – создание образов для загрузки по PXE (не обязательно для Геном);
USE_SSO – настройка интеграции с сервисом аутентификации Keycloak (можно отключить, если авторизация не требуется);
CHANGE_PG_SETTINGS – настройка параметров PostgreSQL (если опция отключена, будут использованы параметры по умолчанию.
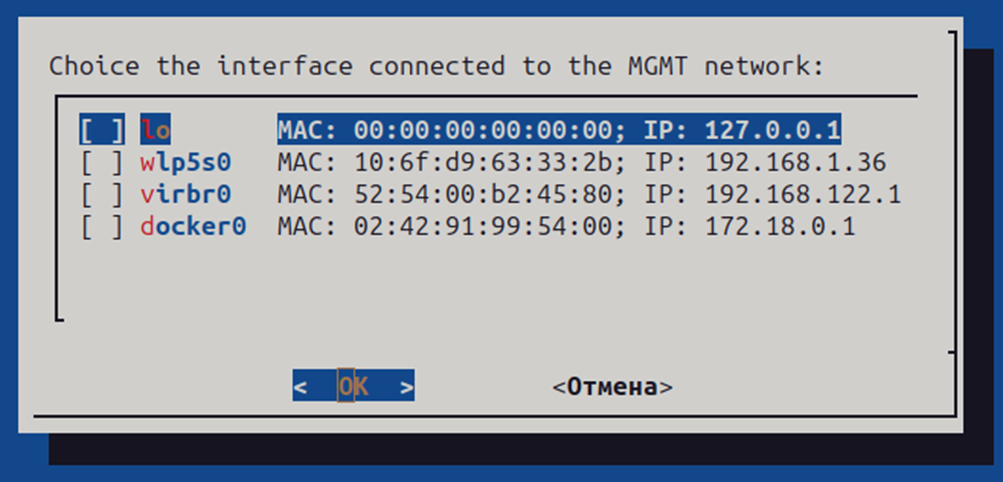
1.4.10 Если выбрана опция USE_DNSMASQ, то отобразится окно выбора интерфейса в сети MGMT:
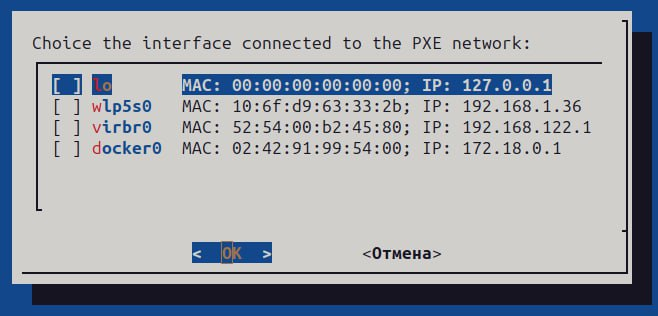
1.4.11 Если выбрана опция CREATE_PXE, то отобразится окно выбора интерфейса в сети PXE:
1.4.12 Выполнить ввод параметров развёртывания:
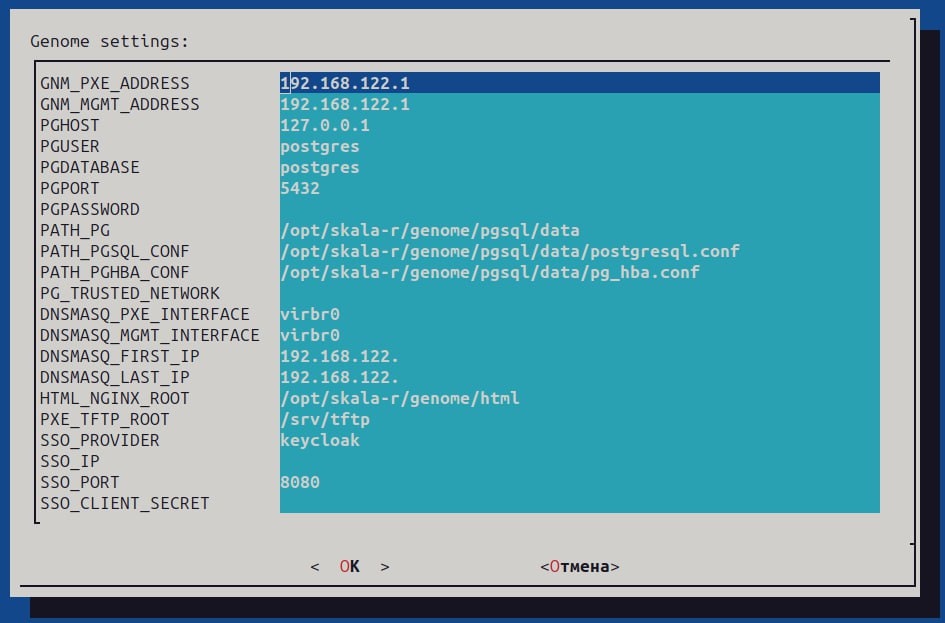
GNM_PXE_ADDRESS – IP-адрес ПО “Скала^р Геном”, предназначенный для первоначальной загрузки хостов будущего кластера через PXE-сеть в случае, если условиями развёртывания предусмотрено наличие отдельной PXE-сети. В случае отсутствия PXE-сети в среде развёртывания, должен совпадать с основным адресом ПО “Скала^р Геном”;
GNM_MGMT_ADDRESS – IP-адрес модуля управления ПО «Скала^р Геном»;
PGHOST – IP-адрес Postgres;
PGUSER – имя пользователя Postgres;
PGDATABASE – имя базы данных Postgres;
PGPORT – порт Postgres;
PGPASSWORD – пароль Postgres;
PATH_PG – путь к файлам Postgres;
PATH_PGSQL_CONF – путь к файлу настроек БД Postgres;
PATH_PGHBA_CONF – путь к файлу настроек доступа к БД Postgres;
PG_TRUSTED_NETWORK – доверенная сеть Postgres (значение поля должно быть записано в формате CIDR, с указанием маски, например 127.0.0.1/32);
DNSMASQ_PXE_INTERFACE – сетевой интерфейс, на котором будут запущены сервисы DHCP, tftp;
DNSMASQ_MGMT_INTERFACE – сетевой интерфейс, на котором будет запущен сервис DNS;
DNSMASQ_FIRST_IP – начальный IP-адрес диапазона DHCP DNSMasq;
DNSMASQ_LAST_IP – конечный IP-адрес диапазона DHCP DNSMasq;
HTML_NGINX_ROOT – путь до корневого каталога html, в котором находятся бинарные артефакты;
PXE_TFTP_ROOT – путь до корневого каталога tftp, из которого раздаются бинарные артефакты при загрузке;
SSO_PROVIDER – поставщик услуг SSO;
SSO_IP – IP-адрес SSO;
SSO_PORT – порт SSO;
SSO_CLIENT_SECRET – секретный ключ SSO.
1.4.13 После ввода всех необходимых данных нажать кнопку <ОК>.
Должен начаться процесс настройки:
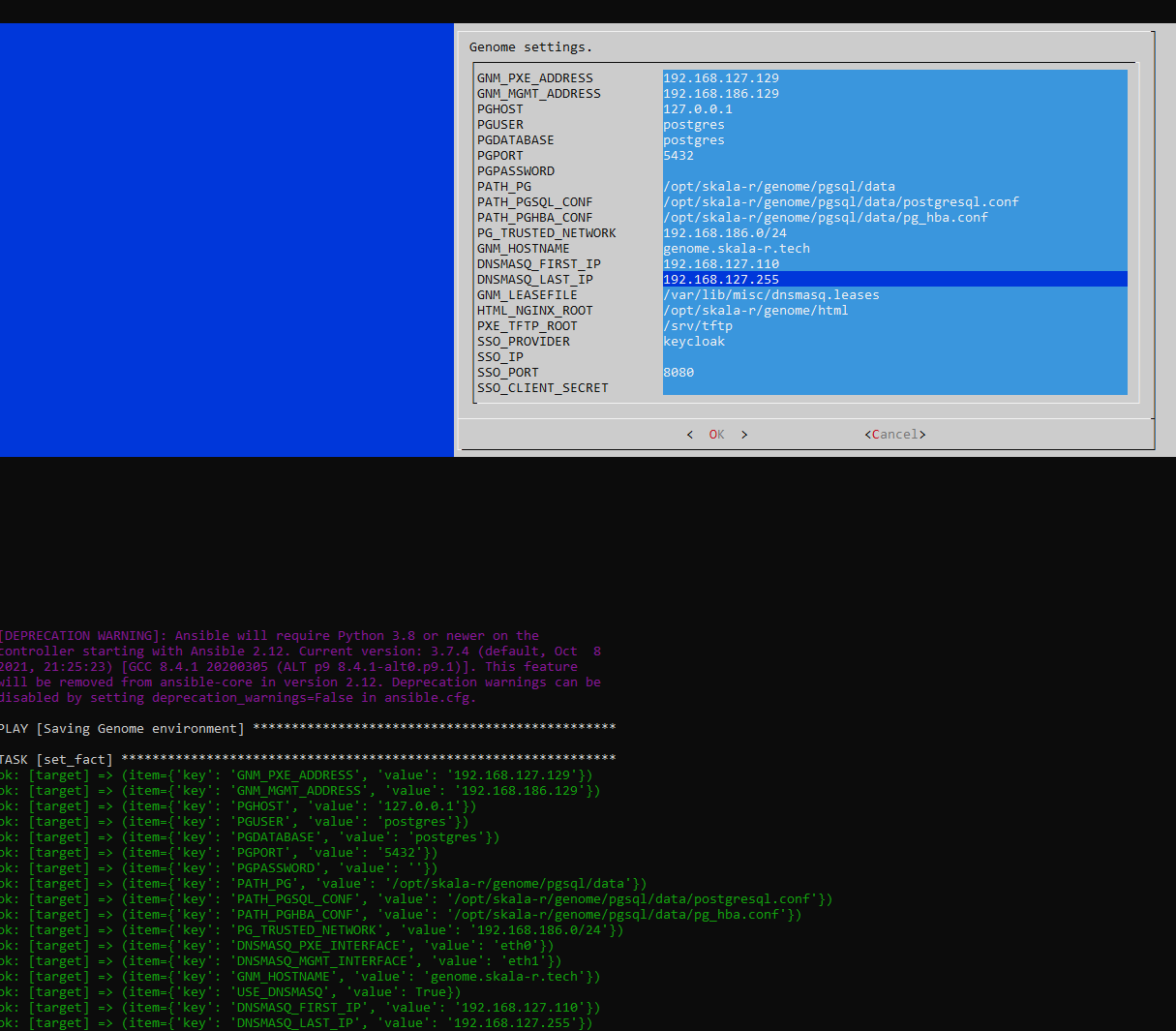
Дождаться завершения процесса настройки:

1.4.14 Перейти в браузер и ввести IP-адрес, установленный в поле GNM_ADDRESS п. 1.4.12 (порт 50888).
Должен открыться интерфейс модуля управления ПО «Скала^р Геном»:
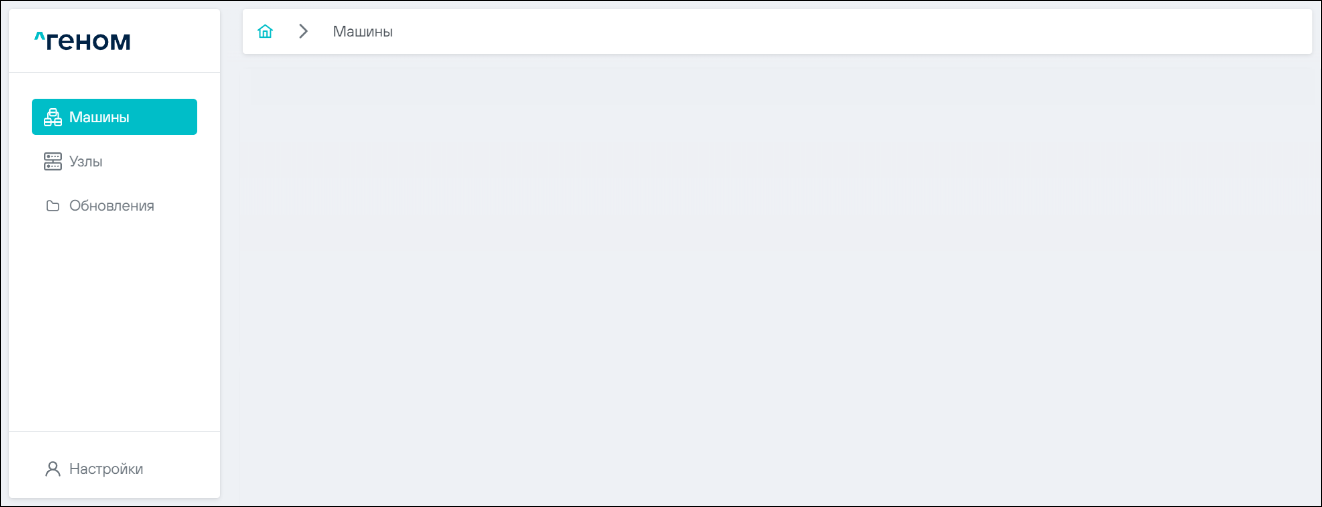
Если в процессе выполнения пп. 1.4.9 и 1.4.12 было установлено использование SSO, то отобразится окно авторизации:
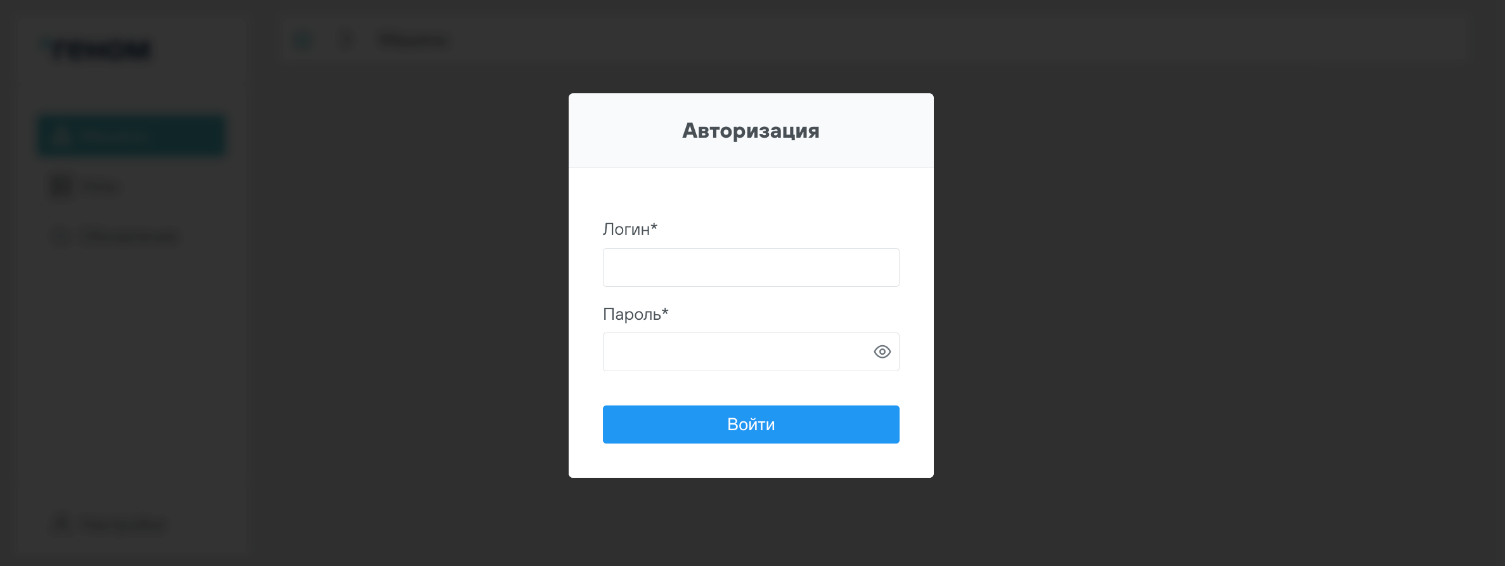
Для доступа к интерфейсу модуля управления ПО “Скала^р Геном” ввести логин, пароль и нажать на кнопку  .
.
Модуль управления “Скала^р Геном” считается успешно установленным, если после отображения интерфейса нет уведомлений об ошибках.
2 Конфигурация#
Конфигурирование модуля управления ПО «Скала^р Геном» производится посредством редактирования файла genome.json.
Назначение полей файла конфигурации genome.json следующее:
| Поле | Значение |
|---|---|
| mp_chroot | Путь для монтирования устройства, куда будет устанавливаться ОС на этапе LiveCD |
| genome_ip | Текущий IP-адрес Геном в сети Управления |
| chroot_env | Строка chroot с установкой необходимых переменных |
| genome_nginx_path | Путь до каталога html, в котором находятся бинарные артефакты |
| mbd_data_request_limit | Лимит запросов (для МБД.П) |
| genome_env | Раздел настройки установки Геном |
| GNM_PXE_ADDRESS | Текущий IP в сети PXE |
| GNM_MGMT_ADDRESS | Текущий IP в сети Управления |
| CHANGE_PG_SETTINGS | Изменение параметров Postgres по умолчанию (true/false) |
| PGHOST | IP-адрес подключения к Postgres Геном |
| PGUSER | Имя пользователя Postgres |
| PGDATABASE | Имя БД Postgres |
| PGPORT | Порт Postgres |
| PGPASSWORD | Пароль пользователя Postgres |
| PATH_PG | Путь к каталогу данных Postgres |
| PATH_PGSQL_CONF | Путь к файлу postgresql.conf |
| PATH_PGHBA_CONF | Путь к файлу pg_hba.conf |
| PG_TRUSTED_NETWORK | Доверенная подсеть Postgres, из которой не требуется ввод пароля |
| DNSMASQ_PXE_INTERFACE | Интерфейс, на котором запущены DHCP и tftp сервисы, предоставляемые DNSMasq |
| DNSMASQ_MGMT_INTERFACE | Интерфейс, на котором запущен DNS сервис, предоставляемый DNSMasq |
| USE_DNSMASQ | Включение и настройка DNSMasq при установке Геном (true/false) |
| DNSMASQ_FIRST_IP | Начальный IP-адрес DHCP пула адресов |
| DNSMASQ_LAST_IP | Конечный IP-адрес DHCP пула адресов |
| CREATE_PXE | Создание образа и настройка PXE (true/false) |
| HTML_NGINX_ROOT | Путь до каталога html, в котором находятся бинарные артефакты |
| PXE_TFTP_ROOT | Путь до каталога tftp |
| USE_SSO | Настройка подключения к SSO сервису (true/false) |
| SSO_PROVIDER | Поставщик услуг SSO |
| SSO_IP | IP-адрес SSO |
| SSO_PORT | Порт SSO |
| SSO_CLIENT_SECRET | Секретный ключ SSO |
3. Журналирование#
3.1. Виды журналов#
В текущей версии реализованы два вида журналов: journal (Лог) и audit (Аудит). На данный момент все записи заносятся в оба журнала.
3.1.1. Лог#
Записи журнала располагаются в директории /opt/skala-r/var/log/genome_mgmt/journal.
Формат журнала - произвольный.
Пример записи:
2024-09-13T18:37:02+0300 user-genome-test INFO Приложение запущено
3.1.2 Аудит#
Записи журнала располагаются в директории /opt/skala-r/var/log/genome_mgmt/audit.
Аудит представлен в форматах CEF и JSON.
3.1.2.1 Формат CEF#
Записи в формате CEF располагаются в директории /opt/skala-r/var/log/genome_mgmt/audit/cef.
Пример записи:
CEF:0|Skala-r|genome_mgmt|1.13|10000040|Запуск: версия Геном.У|3|externalId=2 msg=Приложение запущено deviceProcessName=genome_core outcome=success start=1726241821961 dhost=user-genome-test
Формат CEF имеет обязательные и необязательные поля.
Подстрока с обязательными полями:
CEF:0|Skala-r|genome_mgmt|1.13|20000040|Запуск: версия Геном.У|3|,
где
CEF:0 - версия формата CEF;
Skala-r - название производителя;
genome_mgmt - название продукта;
1.13 - версия продукта;
20000040 - уникальный идентификатор категории события. ID event в таблице ресурсов;
Запуск: версия Геном.У - имя события;
3 - важность события.
Подстрока с расширенными полями:
externalId=2 msg=Приложение запущено deviceProcessName=genome_core outcome=success start=1726241821961 dhost=user-genome-test
Из записей в формате CEF на данный момент выводятся следующие параметры:
externalId - идентификатор события;
msg -подробная информация о событии;
deviceProcessName - имя процесса, ассоциированного с событием;
outcome - результат события;
start - время возникновения события;
dhost - FQDN или имя хоста получателя.
3.1.1.2 Формат JSON#
Записи в формате JSON располагаются в директории /opt/skala-r/var/log/genome_mgmt/audit/json.
Пример записи:
{“createdAt”: “1726241825430”, “userNode”: “yudobrolyubova-genome-test”, “metamodelVersion”: “1”, “module”: “genome_mgmt”, “name”: “Авторизация: Неизвестно (доступ запрещен)”, “params”: [{“name”: “message”, “value”: “Ошибка доступа: 401 url /api/datasheet”}, {“name”: “serviceVersion”, “value”: “1.13”}]}
Требования к формату не указаны.
На данный момент из записей в формате json выводятся следующие параметры:
createdAt - время записи в журнал;
userNode - имя узла;
metamodelVersion - версия модели, на данный момент выставлена в “1”;
module - название приложения;
name - строка с событием;
params - структуры с версией приложения и текстовым сообщением.
3.2 Конфигурационный файл#
Управление записями журналов производится с помощью конфигурационного файла logging_mgmt_conf.yml, расположенного в директории /opt/skala-r/genome/python-modules/lib/python3/site-packages/genome_ng/logging_mgmt_conf.yml.
Пример:
# Все ключи являются необязательными
version: 1
# Аудит
audit:
# Уровень сообщений, которые логируются DEBUG\|INFO\|WARNING\|ERROR\|CRITICAL
level: INFO
# Параметры форматов записи логов
formatters:
# Формат CEF
cef:
# Вкл/выкл запись логов в формате CEF
enable: true
# Управление ротированием файлов логов
rotate:
filepath: "/opt/skala-r/var/log/genome_mgmt/audit/cef/genome_mgmt_audit.log"
maxBytes: 104857600 # максимальный размер файла логов
backupCount: 5 # количество сохраненных логов "<filepath>.<n>" по maxBytes
# Формат JSON (ГосТех)
json:
enable: true
rotate:
filepath: "/opt/skala-r/var/log/genome_mgmt/audit/json/genome_mgmt_audit.log"
maxBytes: 104857600
backupCount: 5
# Фильтрация записей по группам/действиям/статусам событий
# Например, ["11-15", "2"] — исключить с 11 по 15 включительно и 2 ID
# Перечень срезов genome-core/docs/events
filters:
groups: []
actions: []
status: []
# Журналирование, аналогично параметрам аудита
journal:
level: INFO
formatters:
text:
enable: true
rotate:
filepath: "/opt/skala-r/var/log/genome_mgmt/journal/genome_mgmt_journal.log"
maxBytes: 104857600
backupCount: 5
filters:
groups: []
actions: []
status: []
Для включения / выключения какого-либо вида журналов используется параметр enable.
Имена файлов с записями, а также путь до этих файлов, задаются параметром filepath.
Размер файлов и их количество можно указать параметрами maxBytes и backupCount.
Параметром filters производится фильтрация записей в журналах.
Для применения новых настроек после изменения конфигурационного файла необходимо перезапустить приложение.
3.3 Фильтрация записей#
Как было указано, фильтрация записей в журналы осуществляется при помощи параметра filters в конфигурационном файле.
Фильтровать записи возможно по трём параметрам: groups, actions и status.
В коде находится скрипт, генерирующий документацию с описанием этих параметров.
3.3.1 Фильтрация по группам#
Все потенциальные записи разделены на группы в зависимости от типа.
У каждой группы есть имя, описание и идентификатор, по которому можно производить фильтрацию.
Описание параметра groups:
| Атрибут GroupEnum | Имя группы | Текст подгруппы | ID подгруппы |
|---|---|---|---|
| unknown | Неизвестно | Неизвестно | 0 |
| app | Приложение | Приложение genome_mgmt | 10 |
| auth_login | Авторизация | Вход пользователя в систему | 21 |
| auth_logout | Авторизация | Выход пользователя из системы | 22 |
| updates_mgmt | Обновления | Добавление и удаление обновлений в Геном.У | 31 |
| updates_apply | Обновления | Применение обновлений | 32 |
| updates_info | Обновления | Получение информации об обновлениях | 33 |
| updates_internal | Обновления | Внутренняя логика работы с обновлениями | 34 |
| passport_generate | Паспорта | Генерация паспортов | 41 |
| passport_internal | Паспорта | Внутренняя логика работы | 42 |
| node_replace_hw | Замена узла | Регистрация hardware | 51 |
| node_replace_sw | Замена узла | Регистрация software | 52 |
| node_replace_bm | Замена узла | Регистрация benchmark | 53 |
| ipmi_create | IPMI | Создание IPMI | 61 |
| ipmi_check | IPMI | Проверка доступа IPMI | 62 |
| ipmi_info | IPMI | Получение IPMI данных | 63 |
| node_info | Узлы | Получение информации об узлах | 71 |
| node_update | Узлы | Изменение статуса и серийного номера узла | 72 |
| pak_mgmt | ПАК | Регистрация и удаление | 81 |
| pak_info | ПАК | Получение информации об ПАК | 82 |
| cluster_decommissioning | Кластер | Вывод из эксплуатации | 91 |
| cluster_info | Кластер | Получение информации о кластере | 92 |
| cluster_service | Кластер | Работа с сервисами | 93 |
| cluster_pgbouncer | Кластер | Работа с pgbouncer | 94 |
| cluster_uuid | Кластер | Работа с uuid | 95 |
| version | Версия Геном.У | Версия Геном.У | 200 |
3.3.2 Фильтрация по действиям#
Каждой записи соответствует действие, которое зависит от того, что нужно сделать с объектом.
У действий также есть имя и идентификатор, по которому можно фильтровать записи.
Описание параметра actions:
| Атрибут ActionEnum | Имя действия | ID действия |
|---|---|---|
| read | Чтение | 1 |
| update | Редактирование | 2 |
| auth | Авторизация | 3 |
| start | Запуск | 4 |
| stop | Остановка | 5 |
| log_rotate | Ротация логов | 6 |
3.3.3 Фильтрация по статусам#
Каждой записи соответствует статус, обозначающий результат выполнения действия над объектом.
У статусов также есть имя и идентификатор, по которому можно фильтровать записи.
Описание параметра status:
| Атрибут StatusEnum | Текст статуса | ID статуса |
|---|---|---|
| success | Успешно | 0 |
| failed | Неуспешно | 1 |
| forbidden | Доступ запрещен | 2 |
Таким образом, если необходимо, например, исключить все неуспешно завершённые события, в параметр status нужно добавить “1” и перезапустить приложение.
Если необходимо исключить все записи, связанные с заменой узла, в параметр groups нужно добавить “51-53” и перезапустить приложение.
4. Передача файлов журнала во внешние системы#
Для передачи журналов во внешнюю систему используется syslog.
5 Смена пароля служебной БД#
- Подключиться по SSH к машине, на которой установлен Геном. Для этого необходимо запустить консоль/терминал и выполнить команду подключения по SSH:
ssh root@xxx.xxx.xxx.xxx
где xxx.xxx.xxx.xxx – IP-адрес узла, на котором установлен Геном:
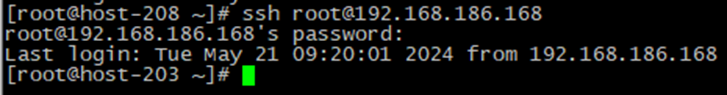
- Подключиться к базе данных пользователем postgres командой:
psql -U postgres
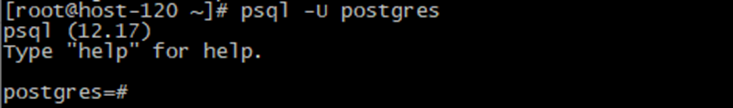
- Сменить пароль на необходимый, выполнив следующий запрос:
ALTER USER postgres with password '1234567890';
где ‘1234567890’ – устанавливаемый пароль:

- Запустить виртуальное окружение следующей командой:
source /opt/skala-r/genome/python-modules/bin/activate

- После запуска виртуального окружения открыть для редактирования файл .vault_store следующей командой:
ansible-vault edit /opt/skala-r/genome/.vault_store

После ввода команды появится уведомление о необходимости ввода пароля доступа к файлу .vault_store. Ввести пароль и нажать Enter.
Пароль доступа к файлу устанавливается на этапе сборки инсталлятора Генома.
- После успешного ввода пароля доступа к файлу .vault_store отобразится окно редактирования доступов:
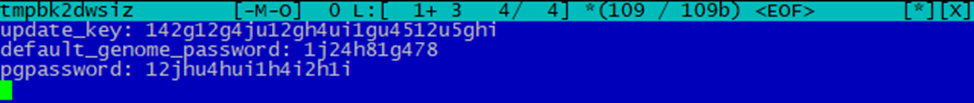
-
Отредактировать пароль к служебной БД, изменив значение после двоеточия в строке pgpassword.
-
Сохранить изменения в файле и выйти из режима редактирования.
-
Выйти из виртуального окружения следующей командой:
deactivate
- Перезапустить сервис genome_ng.service следующей командой:
systemctl restart genome_ng.service
6 Настройка конфигураций систем управления доступом#
6.1 Настройка конфигурации Avanpost#
В конфигурационный файл /opt/skala-r/genome/configuration/genome.json необходимо внести следующие изменения.
В ключе “auth” необходимо указать:
- enabled - установить значение true.
- auth_server - URL Avanpost, например http://fam.skala-r.tech .
- client_id - ID приложения.
- client_secret - значение является ключом из хранилища секретов. Не изменять.
- cert - использование сертификата.
Пример секции авторизации в файле /opt/skala-r/genome/configuration/genome.json:
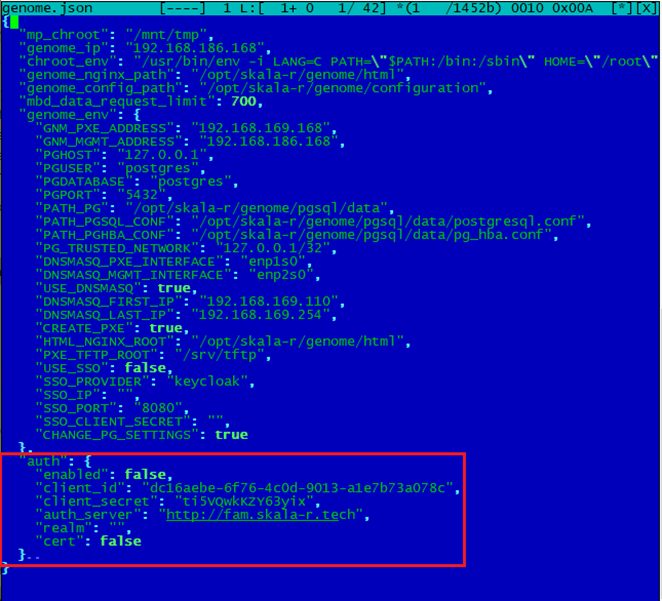
"auth": {
"enabled": true,
"client_id": "dc16aebe-6f76-4c0d-9013-a1e7b73a078c",
"client_secret": "ti5VQwkKZY63yix",
"auth_server": "http://fam.skala-r.tech",
"realm": "",
"cert": false
}
Для применения настроек необходимо перезапустить сервис genome_ng.service командой:
systemctl restart genome_ng.service
Отключение функции регистрации и восстановления пароля
В версии Avanpost FAM v1.10.10.379 отключение Регистрации на странице аутентификации пользователя осуществляется через конфигурационный файл Avanpost, пример пути конфигурационного файла: /opt/idp/config.toml:
[selfregistration]
redirectUrl = 'https://<your_redirect_URL_after_registration>'
disabled = false
Функция “Восстановления пароля” в данной версии отключается через редактирование шаблона страницы аутентификации.
6.2 Настройка конфигурации Keycloak#
В конфигурационный файл /opt/skala-r/genome/configuration/genome.json необходимо внести следующие изменения.
В ключе “auth” необходимо указать:
- enabled - установить значение true.
- auth_server - URL Keycloak, например http://192.168.191.19:8080.
- client_id - ID приложения.
- client_secret - значение является ключом из хранилища секретов. Не изменять.
- realm - realm.
- cert - использование сертификата.
Пример секции авторизации в файле /opt/skala-r/genome/configuration/genome.json:
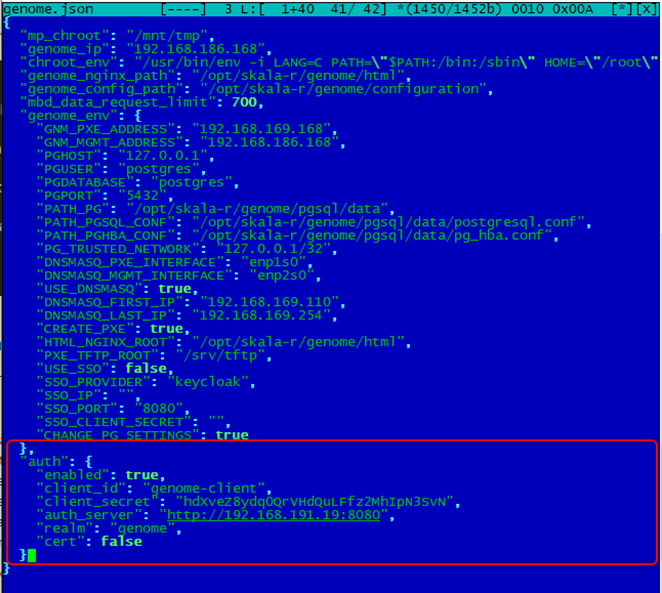
"auth": {
"enabled": true,
"client_id": "genome-client",
"client_secret": "hdXveZ8ydq0QrVHdQuLFfz2MhIpN3SvN",
"auth_server": "http://192.168.191.19:8080",
"realm": "genome",
"cert": false
}