Руководство администратора модуля управления Скала^р Геном версия 1.15#
1 Установка программы#
Для установки и работы ПО «Скала^р Геном» требуется операционная система Альт 8 СП релиз 9, Альт 8 СП релиз 10, Astra Linux Special Edition 1.7.3 (Орёл), RedOS 7.3.
1.1 Минимальные требования к устройству (виртуальной машине):#
- CPU: от 4 ядер;
- RAM: от 16 Гб;
- ROM: от 100 Гб SSD;
- NET: от 1 Гбит/с Ethernet.
1.2 Состав дистрибутива#
Дистрибутив содержит скрипт установки genome-installer.run.
1.3 Используемые сетевые порты#
Порты, необходимы для нормальной работы:
| ПOPT | ПРОТОКОЛ | НАПРАВЛЕНИЕ | НАЗНАЧЕНИЕ |
|---|---|---|---|
| 22 | SSH | I/O | SSH-подключение к модулю управления Геном |
| 5432 | TCP | I/O | PostgreSQL БД модуля управления Геном |
| 48800 | TCP | I/O | REST API модуля управления Геном |
| 50888 | TCP | I/O | Web-интерфейс модуля управления Геном |
| 15000-16000 | HTTP | I/O | IPMI proxies для управления серверами |
Дополнительные порты:
| ПOPT | ПРОТОКОЛ | НАПРАВЛЕНИЕ | НАЗНАЧЕНИЕ |
|---|---|---|---|
| 443 | HTTPS | I/O | Web-интерфейс с подключённым SSL-сертификатом |
1.4 Ход развёртывания#
1.4.1 Открыть терминал.
1.4.2 Проверить тип и версию установленной ОС:
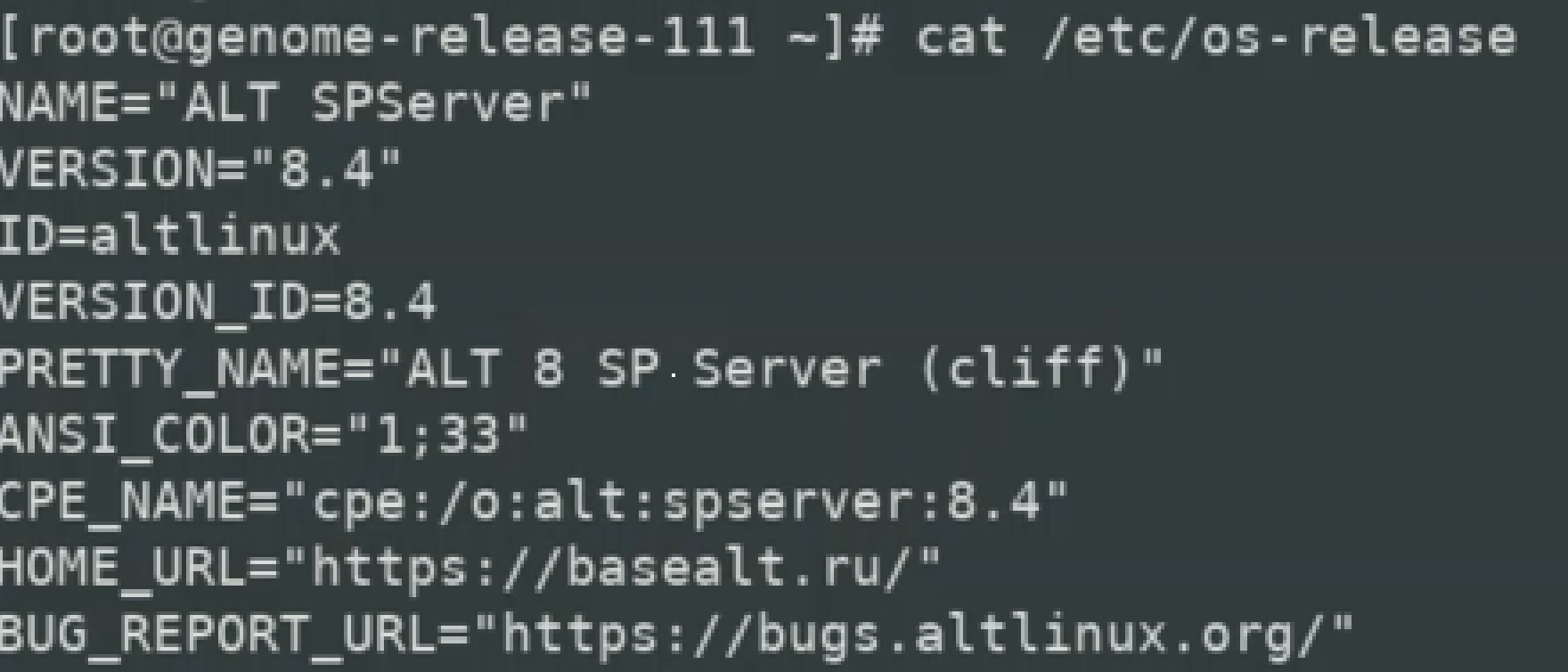
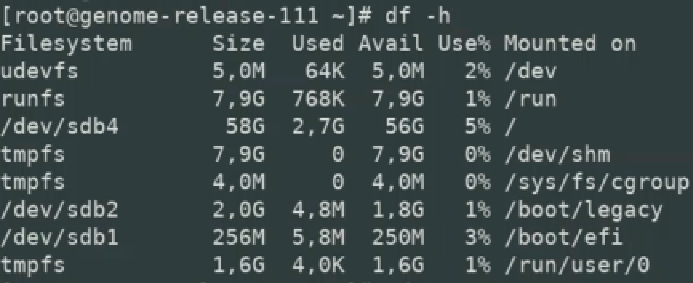
1.4.3 Проверить наличие свободного места для установки модуля управления ПО «Скала^р Геном»:
1.4.4 Перейти в корневой каталог:
1.4.5 Загрузить файл дистрибутива модуля управления ПО «Скала^р Геном» из репозитория.
Дождаться окончания загрузки:

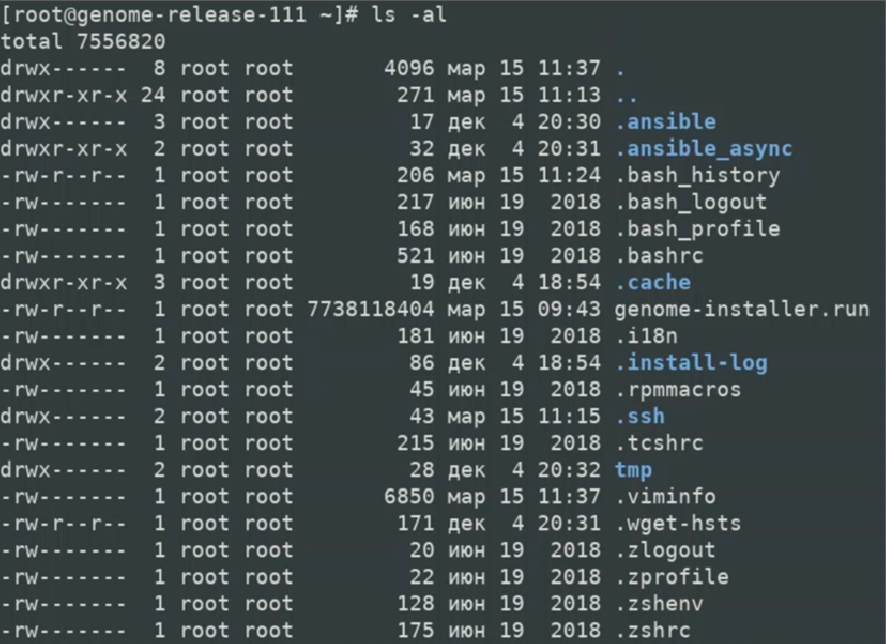
1.4.6 Вывести список файлов в директории и проконтролировать наличие файла genome-installer.run и прав доступа к нему:
1.4.7 Разрешить выполнение файла genome-installer.run:
1.4.8 Запустить выполнение файла genome-installer.run:
Начнётся процесс распаковки и установки модулей:
1.4.9 По завершении выполнения скрипта появится окно выбора опций, в котором выбрать нужную (здесь и далее, в случае необходимости, выбор подтверждается нажатием клавиши ПРОБЕЛ с последующим появлением символа «*» в соответствующем столбце):
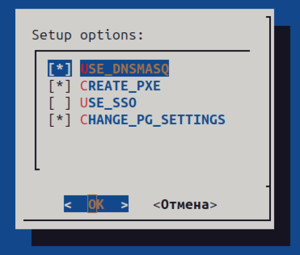
USE_DNSMASQ – настройки сервисов dnsmasq, таких как DNS, DHCP и tftp (не обязательно для Геном);
CREATE_PXE – создание образов для загрузки по PXE (не обязательно для Геном);
USE_SSO – настройка интеграции с сервисом аутентификации Keycloak (можно отключить, если авторизация не требуется);
CHANGE_PG_SETTINGS – настройка параметров PostgreSQL (если опция отключена, будут использованы параметры по умолчанию.
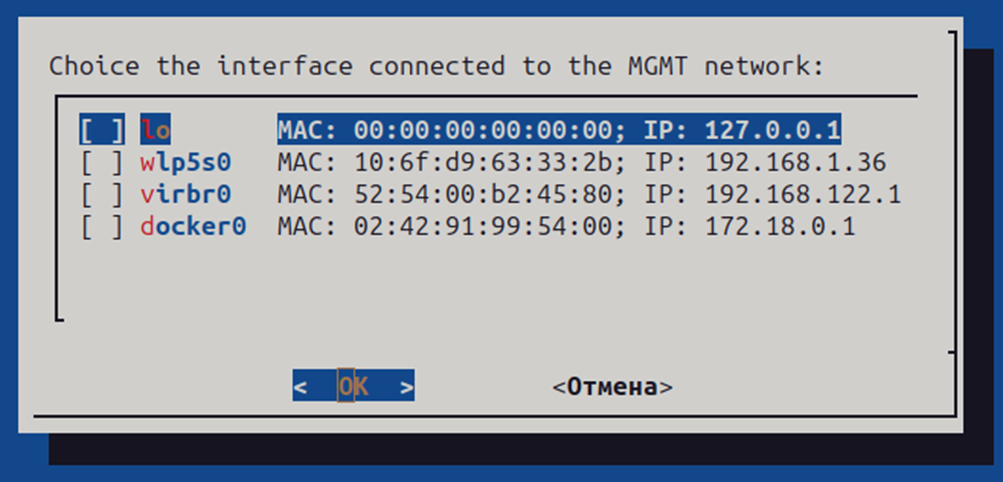
1.4.10 Если выбрана опция USE_DNSMASQ, то отобразится окно выбора интерфейса в сети MGMT:
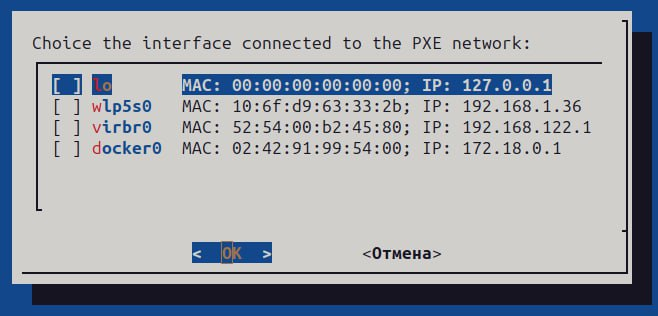
1.4.11 Если выбрана опция CREATE_PXE, то отобразится окно выбора интерфейса в сети PXE:
1.4.12 Выполнить ввод параметров развёртывания:
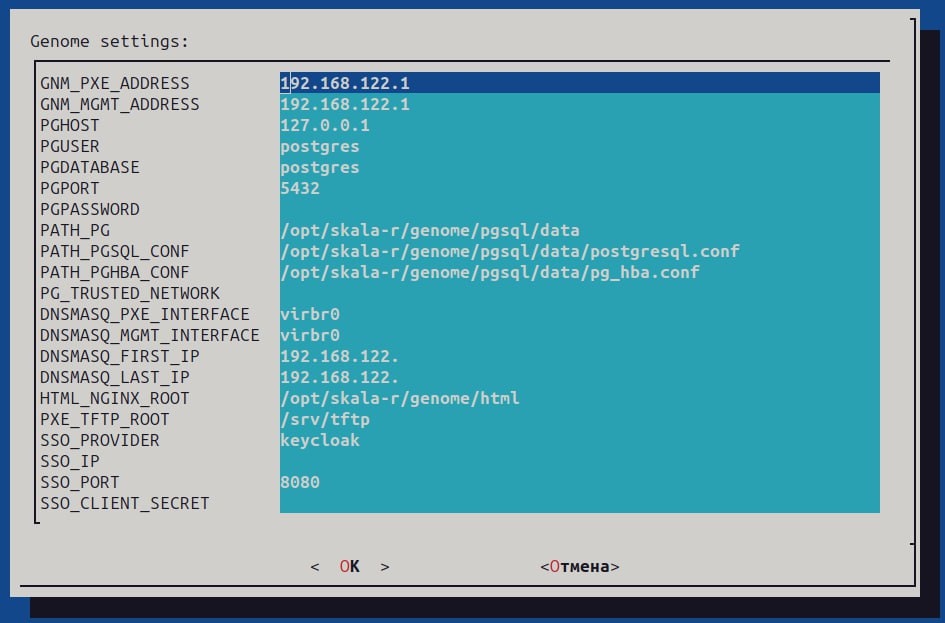
GNM_PXE_ADDRESS – IP-адрес ПО “Скала^р Геном”, предназначенный для первоначальной загрузки хостов будущего кластера через PXE-сеть в случае, если условиями развёртывания предусмотрено наличие отдельной PXE-сети. В случае отсутствия PXE-сети в среде развёртывания, должен совпадать с основным адресом ПО “Скала^р Геном”;
GNM_MGMT_ADDRESS – IP-адрес модуля управления ПО «Скала^р Геном»;
PGHOST – IP-адрес Postgres;
PGUSER – имя пользователя Postgres;
PGDATABASE – имя базы данных Postgres;
PGPORT – порт Postgres;
PGPASSWORD – пароль Postgres;
PATH_PG – путь к файлам Postgres;
PATH_PGSQL_CONF – путь к файлу настроек БД Postgres;
PATH_PGHBA_CONF – путь к файлу настроек доступа к БД Postgres;
PG_TRUSTED_NETWORK – доверенная сеть Postgres (значение поля должно быть записано в формате CIDR, с указанием маски, например 127.0.0.1/32);
DNSMASQ_PXE_INTERFACE – сетевой интерфейс, на котором будут запущены сервисы DHCP, tftp;
DNSMASQ_MGMT_INTERFACE – сетевой интерфейс, на котором будет запущен сервис DNS;
DNSMASQ_FIRST_IP – начальный IP-адрес диапазона DHCP DNSMasq;
DNSMASQ_LAST_IP – конечный IP-адрес диапазона DHCP DNSMasq;
HTML_NGINX_ROOT – путь до корневого каталога html, в котором находятся бинарные артефакты;
PXE_TFTP_ROOT – путь до корневого каталога tftp, из которого раздаются бинарные артефакты при загрузке;
SSO_PROVIDER – поставщик услуг SSO;
SSO_IP – IP-адрес SSO;
SSO_PORT – порт SSO;
SSO_CLIENT_SECRET – секретный ключ SSO.
1.4.13 После ввода всех необходимых данных нажать кнопку <ОК>.
Должен начаться процесс настройки:
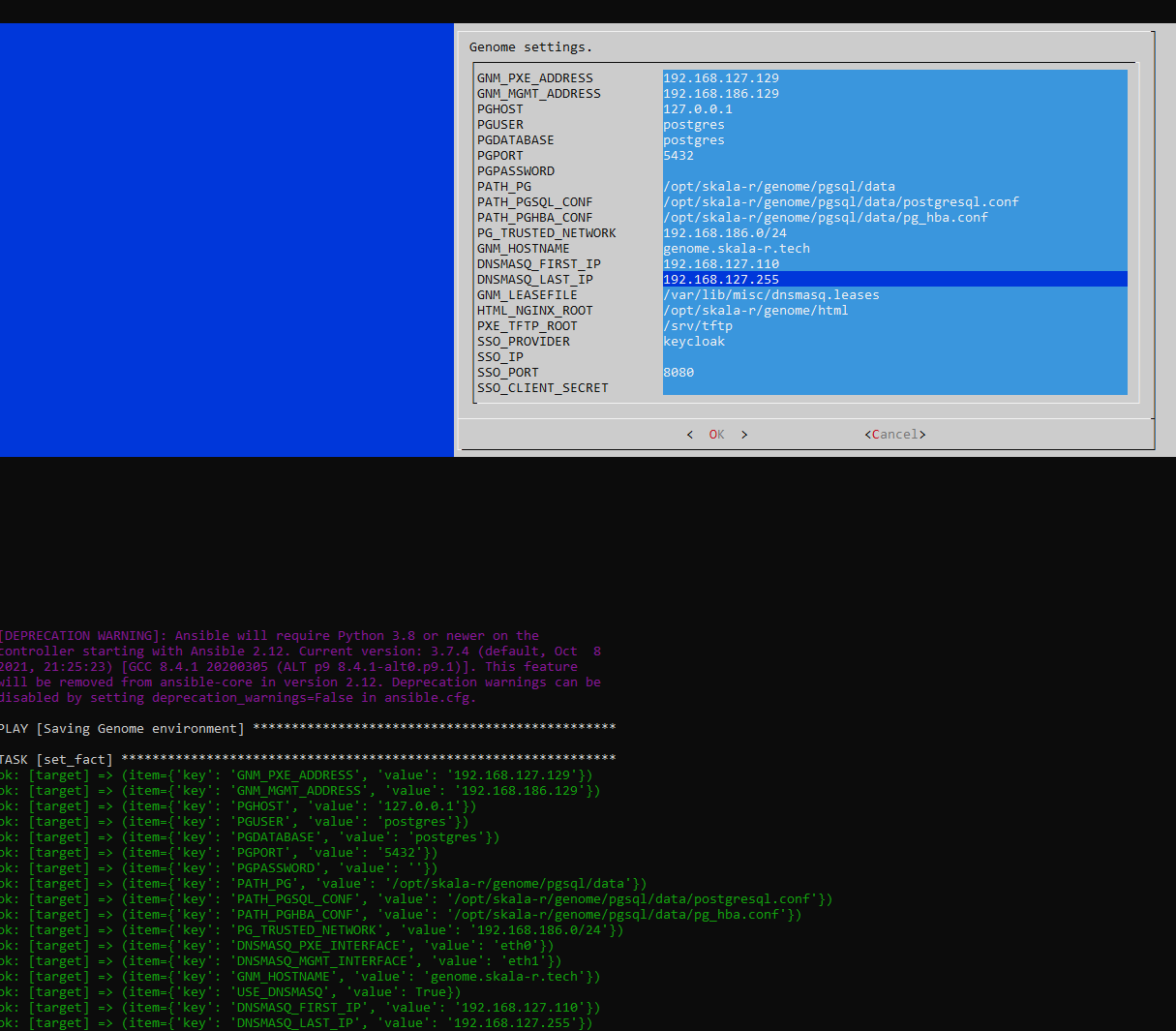
Дождаться завершения процесса настройки:

1.4.14 Перейти в браузер и ввести IP-адрес, установленный в поле GNM_ADDRESS п. 1.4.12 (порт 50888).
Должен открыться интерфейс модуля управления ПО «Скала^р Геном»:
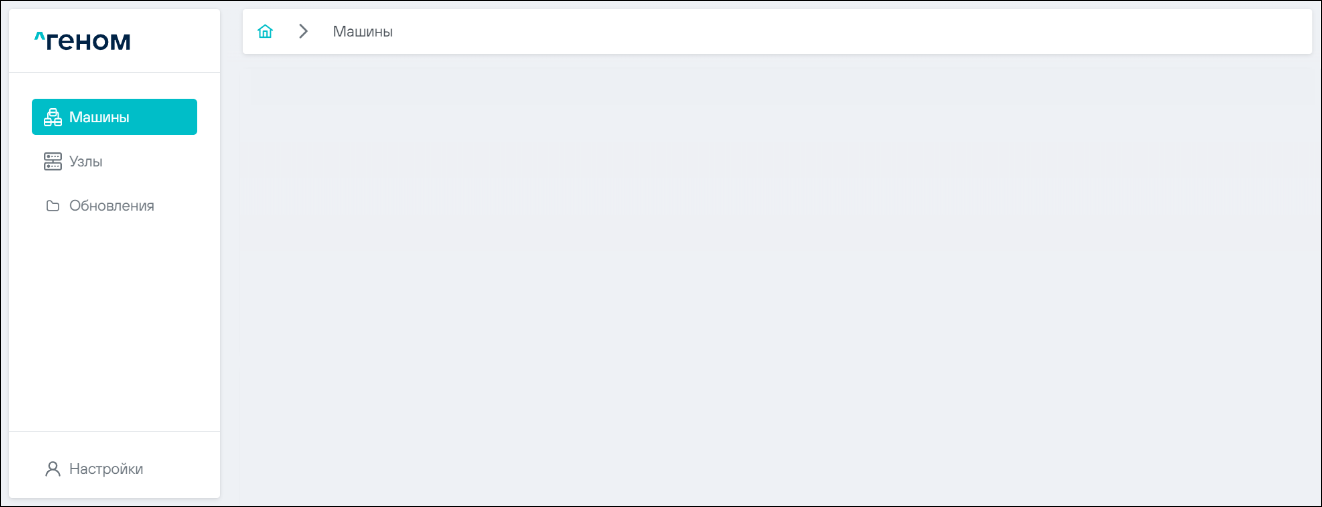
Если в процессе выполнения пп. 1.4.9 и 1.4.12 было установлено использование SSO, то отобразится окно авторизации:
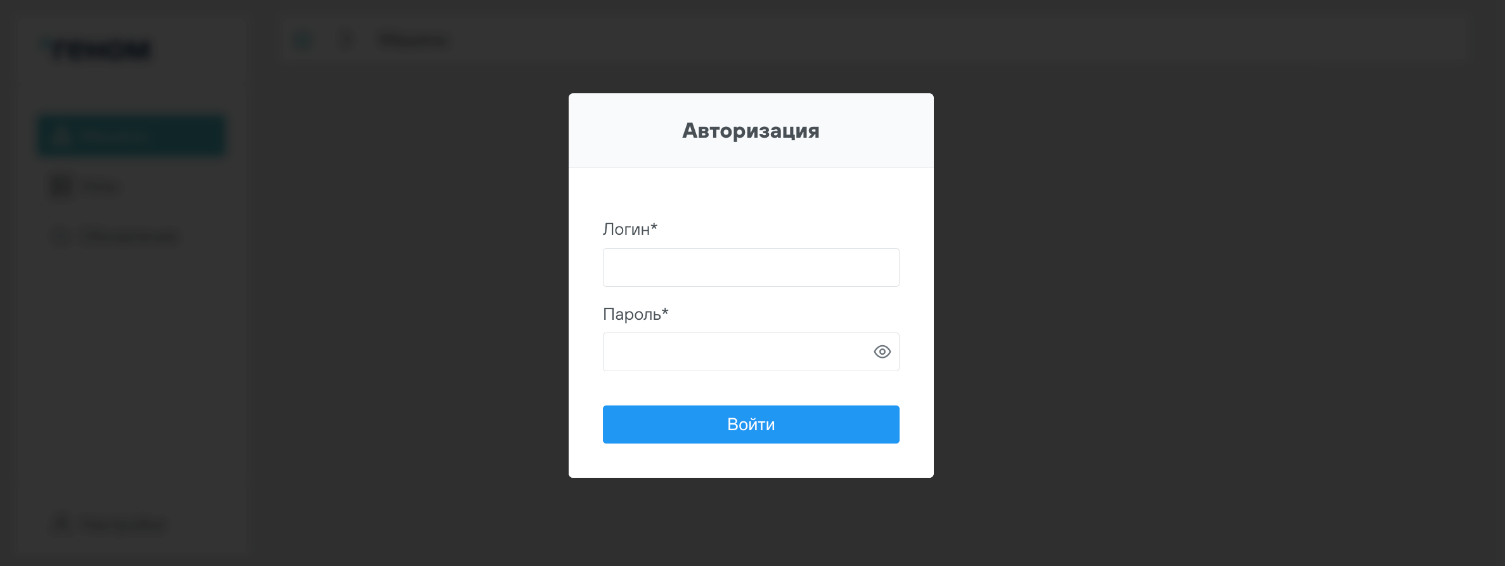
Для доступа к интерфейсу модуля управления ПО “Скала^р Геном” ввести логин, пароль и нажать на кнопку  .
.
Модуль управления “Скала^р Геном” считается успешно установленным, если после отображения интерфейса нет уведомлений об ошибках.
2 Конфигурация#
Конфигурирование модуля управления ПО «Скала^р Геном» производится посредством редактирования файла genome.json.
Назначение полей файла конфигурации genome.json следующее:
| Поле** | Значение |
|---|---|
| mp_chroot | Путь для монтирования устройства, куда будет устанавливаться ОС на этапе LiveCD |
| genome_ip | Текущий IP-адрес Геном в сети Управления |
| chroot_env | Строка chroot с установкой необходимых переменных |
| genome_nginx_path | Путь до каталога html, в котором находятся бинарные артефакты |
| mbd_data_request_limit | Лимит запросов (для МБД.П) |
| genome_env | Раздел настройки установки Геном |
| GNM_PXE_ADDRESS | Текущий IP в сети PXE |
| GNM_MGMT_ADDRESS | Текущий IP в сети Управления |
| CHANGE_PG_SETTINGS | Изменение параметров Postgres по умолчанию (true/false) |
| PGHOST | IP-адрес подключения к Postgres Геном |
| PGUSER | Имя пользователя Postgres |
| PGDATABASE | Имя БД Postgres |
| PGPORT | Порт Postgres |
| PGPASSWORD | Пароль пользователя Postgres |
| PATH_PG | Путь к каталогу данных Postgres |
| PATH_PGSQL_CONF | Путь к файлу postgresql.conf |
| PATH_PGHBA_CONF | Путь к файлу pg_hba.conf |
| PG_TRUSTED_NETWORK | Доверенная подсеть Postgres, из которой не требуется ввод пароля |
| DNSMASQ_PXE_INTERFACE | Интерфейс, на котором запущены DHCP и tftp сервисы, предоставляемые DNSMasq |
| DNSMASQ_MGMT_INTERFACE | Интерфейс, на котором запущен DNS сервис, предоставляемый DNSMasq |
| USE_DNSMASQ | Включение и настройка DNSMasq при установке Геном (true/false) |
| DNSMASQ_FIRST_IP | Начальный IP-адрес DHCP пула адресов |
| DNSMASQ_LAST_IP | Конечный IP-адрес DHCP пула адресов |
| CREATE_PXE | Создание образа и настройка PXE (true/false) |
| HTML_NGINX_ROOT | Путь до каталога html, в котором находятся бинарные артефакты |
| PXE_TFTP_ROOT | Путь до каталога tftp |
| USE_SSO | Настройка подключения к SSO сервису (true/false) |
| SSO_PROVIDER | Поставщик услуг SSO |
| SSO_IP | IP-адрес SSO |
| SSO_PORT | Порт SSO |
| SSO_CLIENT_SECRET | Секретный ключ SSO |
3 Логирование#
Команда journalctl -u genome_ng показывает логи модуля управления ПО «Скала^р Геном» на уровне DEBUG.
В каталоге /opt/skala-r/genome/logs находятся логи с ротацией приложения, в том числе там находится каталог sessions (/opt/skala-r/genome/logs/sessions), в котором находятся логи развёртываний по id сессии.
4 Смена пароля служебной БД#
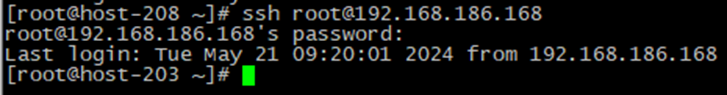
- Подключиться по SSH к машине, на которой установлен Геном. Для этого необходимо запустить консоль/терминал и выполнить команду подключения по SSH:
ssh root@xxx.xxx.xxx.xxx
где xxx.xxx.xxx.xxx – IP-адрес узла, на котором установлен Геном:
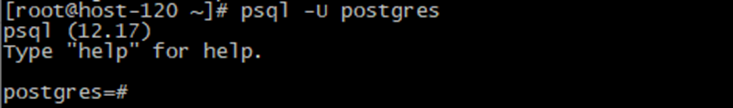
- Подключиться к базе данных пользователем postgres командой:
psql -U postgres
- Сменить пароль на необходимый, выполнив следующий запрос:
ALTER USER postgres with password '1234567890';
где ‘1234567890’ – устанавливаемый пароль:

- Запустить виртуальное окружение следующей командой:
source /opt/skala-r/genome/python-modules/bin/activate

- После запуска виртуального окружения открыть для редактирования файл .vault_store следующей командой:
ansible-vault edit /opt/skala-r/genome/.vault_store

После ввода команды появится уведомление о необходимости ввода пароля доступа к файлу .vault_store. Ввести пароль и нажать Enter.
Пароль доступа к файлу устанавливается на этапе сборки инсталлятора Генома.
- После успешного ввода пароля доступа к файлу .vault_store отобразится окно редактирования доступов:
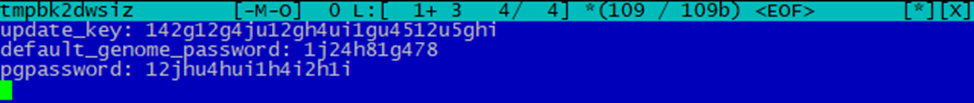
-
Отредактировать пароль к служебной БД, изменив значение после двоеточия в строке pgpassword.
-
Сохранить изменения в файле и выйти из режима редактирования.
-
Выйти из виртуального окружения следующей командой:
deactivate
- Перезапустить сервис genome_ng.service следующей командой:
systemctl restart genome_ng.service